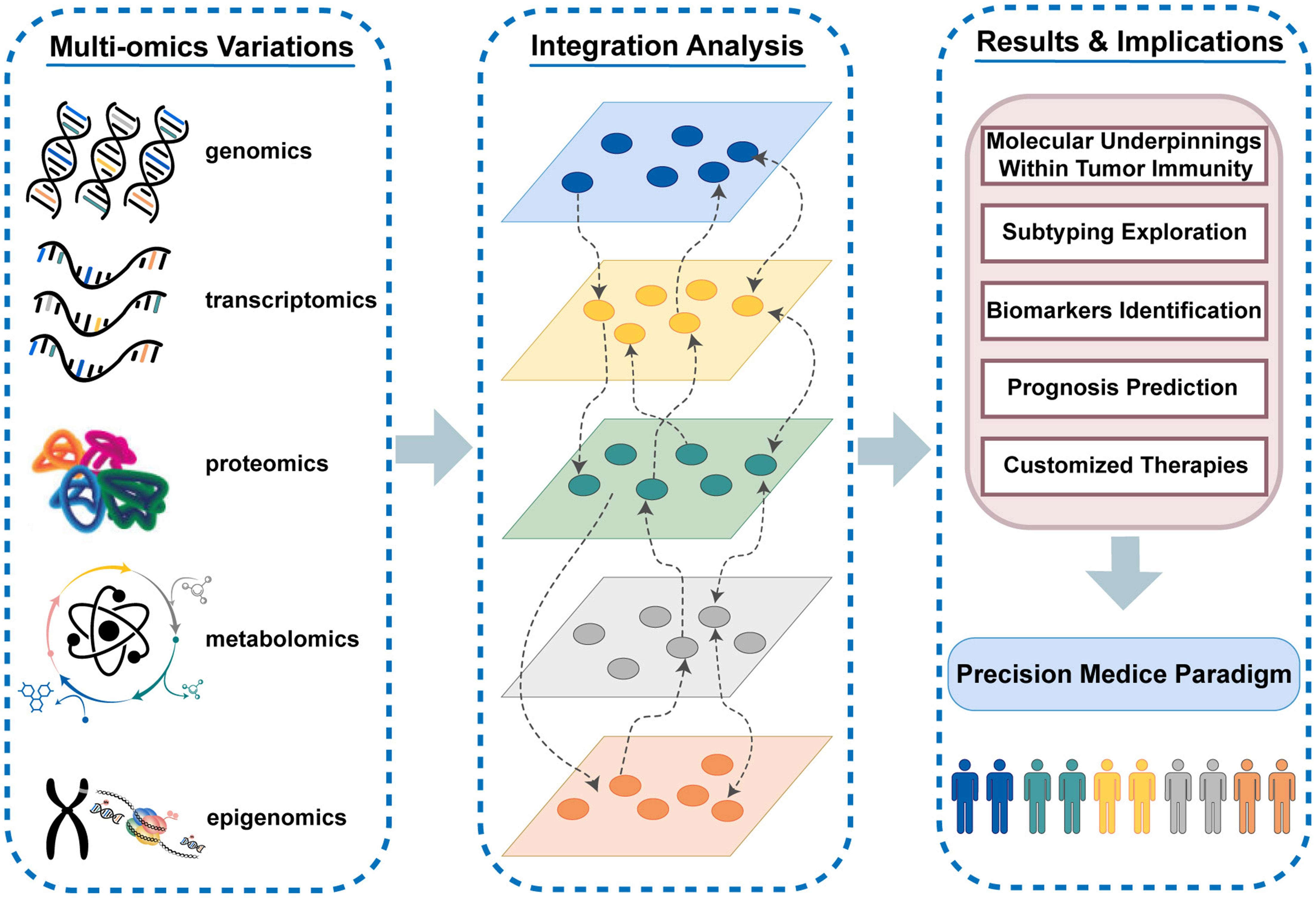
Le terme "omics" désigne un ensemble de disciplines scientifiques qui visent à analyser de manière globale et simultanée les différentes couches de la biologie moléculaire. Ces approches incluent notamment la génomique, la transcriptomique, la protéomique, la métabolomique et l’épigénomique. Elles permettent une compréhension fine des systèmes biologiques, grâce à l'intégration de données massives et multidimensionnelles.
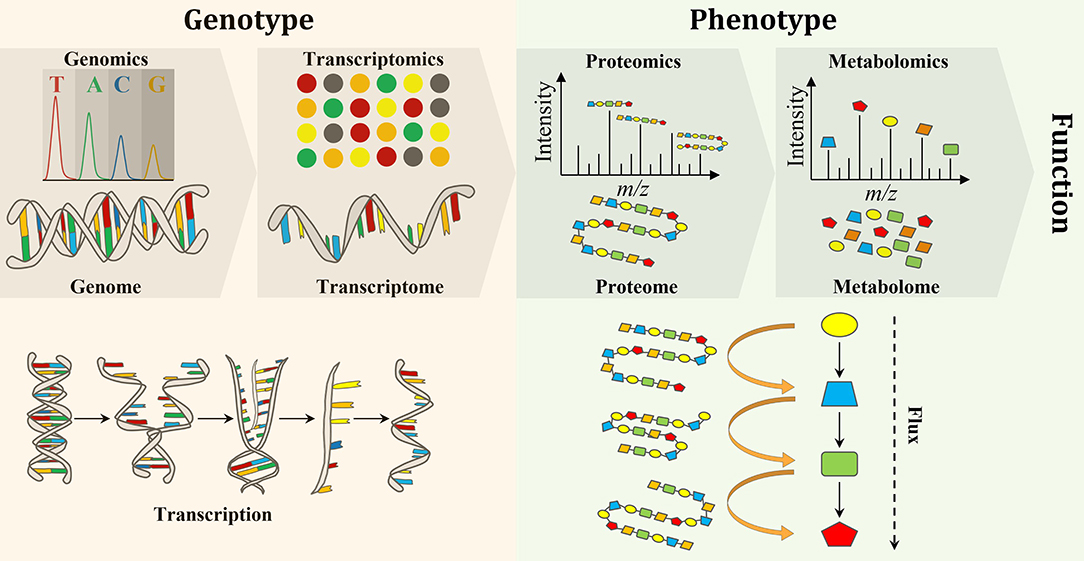
🧬 Génomique : Décoder l’ADN à Grande Échelle
La génomique étudie l’intégralité du génome d’un organisme. Grâce à des techniques comme le séquençage haut débit (NGS), le séquençage complet du génome ou du transcriptome, les chercheurs peuvent :
02

Explorer les gènes régulateurs
01

Identifier des variations génétiques (SNP, CNV)

03

Analyser la structure et l’organisation des chromosomes
04

Étudier la diversité génomique inter-espèces
Transcriptomique : Le Langage de l’ARN
La transcriptomique s’intéresse à l’ensemble des transcrits d’ARN produits dans une cellule à un moment donné. À l’aide de techniques comme le RNA-seq ou le single-cell RNA-seq (scRNA-seq), elle permet :

D’identifier les gènes exprimés

De détecter les ARN non codants (miRNA, lncRNA)

D’observer la réponse transcriptionnelle à divers stimuli

D’explorer la spécificité cellulaire
Protéomique : Cartographier les Protéines Fonctionnelles
La protéomique est la science qui étudie l’ensemble des protéines exprimées par un génome dans un contexte donné. Contrairement à la génomique qui explore des séquences fixes, la protéomique reflète l’état dynamique et fonctionnel des cellules, selon le temps, le type cellulaire ou les conditions environnementales.
Elle permet de comprendre comment les protéines interagissent, se modifient et conduisent les processus biologiques. Cette approche est essentielle pour décoder les fonctions cellulaires complexes et identifier des cibles pour l’ingénierie biomoléculaire.

Méthodes principales utilisées en protéomique

1. Spectrométrie de masse (MS)
La spectrométrie de masse est l’outil central en protéomique. Elle permet d’identifier et de quantifier les protéines présentes dans un échantillon complexe, après digestion enzymatique (souvent par la trypsine).
Les techniques associées incluent :
- LC-MS/MS (chromatographie liquide couplée à la MS) : pour la séparation et l’analyse de peptides.
- MALDI-TOF : pour des analyses rapides de mélanges protéiques.
- iTRAQ / TMT : pour une quantification comparative multi-échantillons par marquage isotopique.

2. Modifications post-traductionnelles (PTMs)
Les PTMs jouent un rôle clé dans la régulation de l’activité protéique. Elles incluent des modifications comme :
- Phosphorylation
- Acétylation
- Ubiquitination
- Glycosylation
- Méthylation
L’analyse fine des PTMs permet de comprendre les mécanismes de signalisation cellulaire, la réponse au stress et la régulation des fonctions enzymatiques.

3. Interactions protéine-protéine (PPI)
Les protéines ne fonctionnent pas de manière isolée. Elles s’associent pour former des complexes multiprotéiques qui réalisent des fonctions précises. La cartographie des réseaux d’interactions (interactome) est essentielle pour :
- Identifier les partenaires fonctionnels d'une protéine cible
- Comprendre les réseaux de signalisation intracellulaires
- Étudier les effets de mutations ou de perturbations environnementales
✅ Outils : co-immunoprécipitation (Co-IP), double hybride, FRET/BRET, crosslinking-MS, pull-down assays

4. Voies de signalisation et dynamique fonctionnelle
La protéomique dynamique permet de suivre en temps réel les variations de l’expression protéique, leur localisation subcellulaire et leur activation fonctionnelle. Ces données sont cruciales pour :
- Décoder les voies de transduction du signal (MAPK, PI3K, JAK/STAT…)
- Étudier les cycles cellulaires et les transitions métaboliques
- Explorer les réseaux de rétroaction dans les systèmes biologiques
✅ Approches utilisées : time-course proteomics, protéomique quantitative, marquage isotopique stable (SILAC)

Métabolomique : Le Profil Biochimique Global
La métabolomique est l’analyse globale des métabolites, ces petites molécules (<1500 Da) issues de l’activité cellulaire. Elle offre une vision directe du phénotype fonctionnel d’un système biologique, complémentaire à la génomique et à la protéomique.

Dresser des profils métaboliques complets
La métabolomique permet de générer une empreinte biochimique représentative de l’état métabolique d’un échantillon, qu’il s’agisse de cellules, de tissus, de milieux extracellulaires ou de biofluides. Ces profils globaux sont obtenus par des approches non ciblées (untargeted metabolomics) qui révèlent des centaines de composés simultanément, sans a priori.
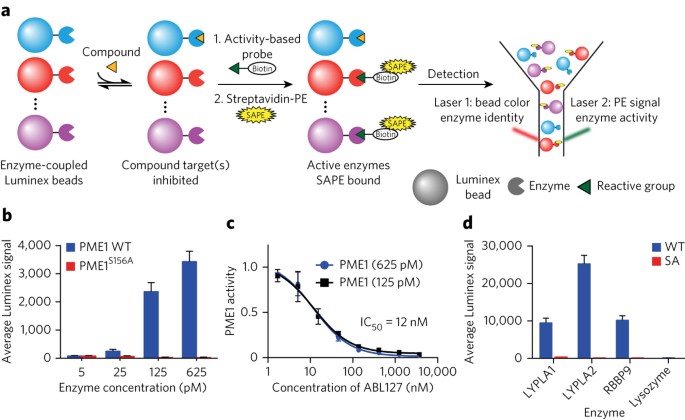
Suivre l’activité enzymatique dans un système
L’analyse métabolomique permet de mesurer les fluctuations de substrats et produits enzymatiques, révélant ainsi l’activité catalytique des enzymes au sein de voies spécifiques. En suivant les variations de concentration en fonction du temps ou des conditions expérimentales, on peut déduire l’efficacité ou l’inhibition de certains enzymes.

Détecter des variations biochimiques
Les technologies métabolomiques permettent de repérer des changements subtils dans la composition chimique interne d’un système, révélateurs de perturbations, de stress, ou d’adaptations. Ces biomarqueurs métaboliques peuvent être quantifiés de manière très sensible grâce à la haute résolution des spectromètres.

Épigénomique : L’Information au-delà du Code Génétique
L’épigénomique étudie l’ensemble des modifications héréditaires de l’expression des gènes qui ne s’accompagnent d’aucun changement dans la séquence d’ADN. Elle révèle comment l’environnement, les signaux biochimiques et l'organisation de la chromatine influencent durablement l'activité génomique.

La méthylation de l’ADN
La méthylation consiste en l’ajout de groupements méthyle sur les cytosines, principalement dans les îlots CpG, entraînant généralement une répression transcriptionnelle. Ce mécanisme est crucial dans le contrôle de l’expression génique, l’empreinte génomique, la différenciation cellulaire et la stabilité chromosomique.

L’accessibilité de la chromatine
L’accessibilité de la chromatine désigne la facilité avec laquelle les protéines régulatrices peuvent interagir avec l’ADN. Des régions ouvertes (euchromatine) sont généralement associées à une transcription active, tandis que des régions condensées (hétérochromatine) sont transcriptionnellement silencieuses.
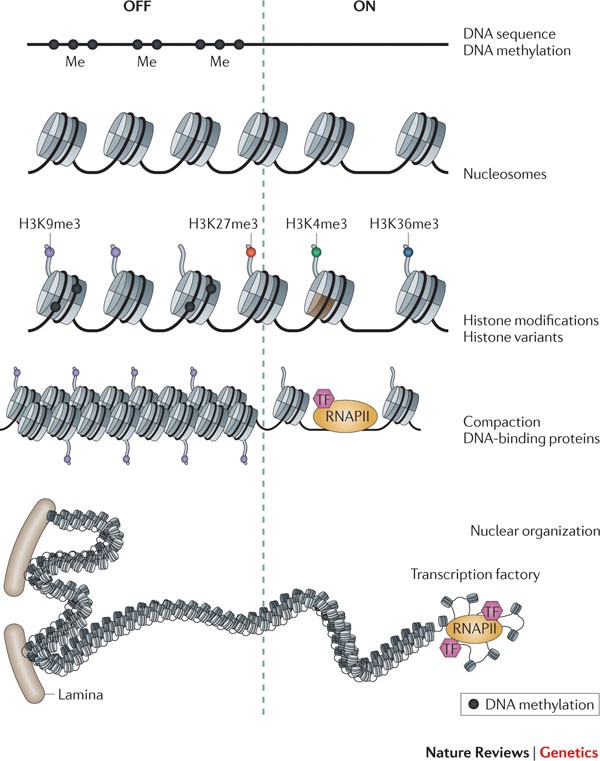
Les modifications des histones
Les histones, protéines autour desquelles l’ADN est enroulé, peuvent subir des modifications (acétylation, méthylation, ubiquitination) qui influencent la compaction de la chromatine. Ces modifications agissent comme un code épigénétique qui active ou inhibe la transcription selon leur combinaison et leur localisation.

Les ARN régulateurs
Les ARN non codants comme les miRNA, lncRNA ou siRNA participent à la régulation post-transcriptionnelle et transcriptionnelle des gènes. Ils peuvent interagir avec l’ADN, les ARN messagers ou les protéines pour moduler l'expression d’un grand nombre de cibles de façon fine et contextuelle.
🔗 Reconstituer des réseaux moléculaires complexes
En croisant les données issues de plusieurs niveaux d’analyse, on peut modéliser les interactions entre gènes, protéines, ARN et métabolites au sein de réseaux dynamiques. Cela permet d’identifier des modules fonctionnels, des nœuds de régulation, ou des interconnexions critiques dans les processus biologiques.

🧬 Décrypter la diversité fonctionnelle entre cellules ou échantillons
La multi-omics permet de comparer des profils biologiques complexes entre différents types cellulaires, conditions expérimentales ou origines biologiques. Elle révèle les signatures moléculaires spécifiques, la variabilité inter-individuelle ou les adaptations environnementales intégrées à plusieurs niveaux.

⚙️ Optimiser l’ingénierie des systèmes biologiques
Grâce à une compréhension intégrée des flux d'information du gène au métabolite, la multi-omics facilite la modélisation rationnelle et l’amélioration de circuits biologiques dans les domaines comme la biologie synthétique, la bio-ingénierie ou la production biotechnologique. Cette approche favorise l’optimisation des voies métaboliques, la réécriture génomique ciblée et la prévision des comportements cellulaires.

🤖 Générer des modèles prédictifs à l’aide de l’intelligence artificielle
Les jeux de données omiques multidimensionnels sont idéaux pour entraîner des algorithmes d’apprentissage automatique capables de détecter des corrélations cachées, de prédire des comportements biologiques ou de classer des échantillons complexes. L’intelligence artificielle appliquée à la biologie offre ainsi des outils décisionnels puissants pour l’exploration de systèmes vivants à grande échelle.

📈 Vers une biologie des systèmes intégrative
La biologie multi-omics constitue aujourd’hui un levier incontournable pour la recherche en sciences du vivant computationnelles, en analyse de données biologiques, et en modélisation intégrée. En connectant les différentes couches d'information moléculaire, elle permet une reconstruction fidèle de la logique interne des systèmes vivants, ouvrant la voie à une exploration prédictive, personnalisée et rationalisée de la biologie.

Exemple multi-omics – Blé (Triticum aestivum)
Objectif :
Améliorer le rendement du blé en conditions de stress environnemental (sécheresse, chaleur) en identifiant des signatures moléculaires intégrées.
Approche multi-omics :
- Génomique : Identification de variants génétiques liés à la tolérance.
- Transcriptomique : Analyse de gènes exprimés sous stress hydrique.
- Protéomique : Détection de protéines protectrices (HSP, enzymes antioxydantes).
- Métabolomique : Profilage de métabolites liés à la résilience (proline, tréhalose).
- Épigénomique : Observation de changements de méthylation affectant l’expression génique.
Résultat :
Intégration des données a permis d’identifier un module moléculaire clé corrélé au rendement. Utilisé pour sélectionner des lignées élites avec +22 % de biomasse en condition de stress.
🧠 Outils Bioinformatiques et Analyse des Données Omics
Les approches omiques produisent des volumes massifs de données, analysés à l’aide de solutions spécialisées :
- R, Python, Bioconductor, Galaxy
- Bases de données ouvertes (Ensembl, GEO, UniProt)
- Plateformes cloud pour l’analyse distribuée
- Visualisation interactive (Shiny, Jupyter, Dash)

📌 Conclusion : Pourquoi les Omics Sont Incontournables
Les technologies "omics" ont profondément transformé les sciences de la vie. Elles permettent une exploration exhaustive des mécanismes biologiques, depuis la séquence génétique jusqu’aux processus métaboliques.
👉 L'intégration des données omiques est aujourd’hui un pilier incontournable de la biotechnologie, de la génomique fonctionnelle, de l’ingénierie cellulaire, de la microbiologie environnementale, et bien plus encore.
Omics technologies are revolutionizing life science by enabling large-scale analysis of genes, proteins, and metabolites, and their integration with advanced tools like CellTiter-Glo assays enhances cellular viability studies across culture medium formulations. Researchers now combine omics omics approaches with precision materials such as PLGA polymer, DSPA, and Jeffamine to build nanocarriers using lipids like ALC-0315, ALC-0159, and DDAB. These systems often incorporate cyclodextrin or strep-tag II for targeted delivery and rely on visualization dyes like Acridine Orange and Nile Red. Thermal processing equipment is key in preparing nanoparticles with precise 1um sizing, while reagents like malic acid (Deutsch) and sodium citrate (Svenska) are optimized for stability. In protein analysis, protein ladders and Stains-All provide accurate separation, and tools such as PEI transfection and Hemasure support high-throughput workflows. Whether scaling up with 2.5x10 batch sizes or ensuring regulatory-grade quality in SSA Medizin applications, omics-driven research continues to advance biomedical innovation.